

Descoberta realizada a partir da análise de amostras de tecido e de saliva de pacientes com câncer de boca mostra correlação de uma assinatura de três peptídeos com a presença de metástase (imagem: Avaliação da abundância de proteínas selecionadas em amostras independentes de tecido de pacientes utilizando anticorpos por meio da imuno-histoquímica/ Nature Communications)
Descoberta realizada a partir da análise de amostras de tecido e de saliva de pacientes com câncer de boca mostra correlação de uma assinatura de três peptídeos com a presença de metástase
Descoberta realizada a partir da análise de amostras de tecido e de saliva de pacientes com câncer de boca mostra correlação de uma assinatura de três peptídeos com a presença de metástase
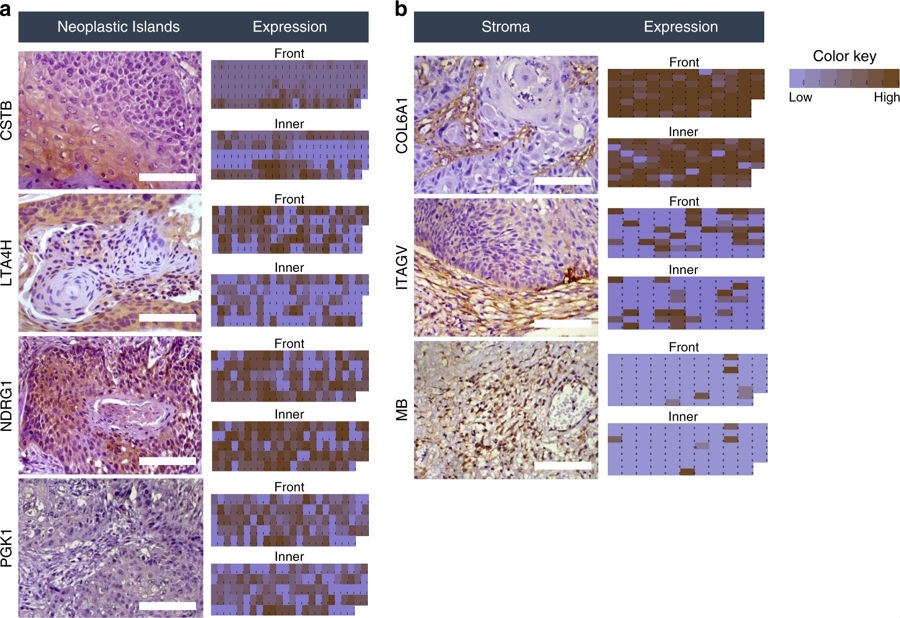
Descoberta realizada a partir da análise de amostras de tecido e de saliva de pacientes com câncer de boca mostra correlação de uma assinatura de três peptídeos com a presença de metástase (imagem: Avaliação da abundância de proteínas selecionadas em amostras independentes de tecido de pacientes utilizando anticorpos por meio da imuno-histoquímica/ Nature Communications)
Maria Fernanda Ziegler | Agência FAPESP – Pesquisadores identificaram a correlação entre a abundância de proteínas presentes no tecido tumoral e na saliva com a progressão do câncer de boca.
A descoberta surge como um parâmetro capaz de antecipar ou prever a progressão da doença – se há a presença ou ausência de metástase em linfonodo cervical, por exemplo –, além de superar as limitações dos exames clínicos e de imagem utilizados na clínica e orientar a escolha do tipo de tratamento ideal para cada paciente.
O estudo se iniciou na fase de descoberta por meio da análise proteômica de diferentes áreas do tecido tumoral utilizando-se 120 amostras microdissecadas e na fase de verificação as assinaturas de prognóstico foram confirmadas em aproximadamente 800 amostras de tecido por meio da técnica de imuno-histoquímica – localização de antígenos em tecidos, explorando o princípio da ligação específica de anticorpos a antígenos no tecido biológico – e 120 amostras de saliva de pacientes com a doença por proteômica baseada em alvos ou dirigida.
“O conjunto de dados nos levou a ter um resultado robusto e bastante promissor na definição da gravidade da doença. Além de sugerirmos marcadores potenciais da doença em uma primeira fase, também verificamos esses marcadores em uma segunda fase da pesquisa, o que confere mais confiabilidade aos achados, mostrando que esses marcadores são eficientes para classificar o paciente com metástase em linfonodo cervical”, disse Adriana Franco Paes Leme, pesquisadora do Laboratório Nacional de Biociências (LNBio), no Centro Nacional de Pesquisas em Energia e Materiais (CNPEM), e autora correspondente de artigo publicado na Nature Communications sobre o estudo.
O trabalho, apoiado pela FAPESP, foi conduzido no CNPEM em parceria com o Instituto do Câncer do Estado de São Paulo (Icesp) e a Faculdade de Odontologia de Piracicaba da Universidade Estadual de Campinas (FOP-Unicamp), o Instituto de Ciências Matemáticas e de Computação da Universidade de São Paulo (USP), o Instituto de Computação da Universidade Estadual de Campinas e a Faculdade de Odontologia da Universidade Estadual do Oeste do Paraná, entre outras instituições nacionais e internacionais.
O câncer de boca, também chamado de carcinoma espinocelular (CEC), é o tipo mais comum de tumor maligno de cabeça e pescoço. Tem alta prevalência e mortalidade, com cerca de 300 mil novos casos diagnosticados por ano no mundo e 145 mil mortes. Embora seja relativamente fácil de ser detectado, por feridas na boca identificadas por dentistas, geralmente o diagnóstico é feito quando a doença já está em estágio avançado.
“O estudo levou cinco anos até chegarmos a essa descoberta. Foi dividido em duas fases. Na primeira, usamos a proteômica baseada em descoberta, quando identificamos e quantificamos as proteínas dos tecidos tumorais. Na segunda fase do estudo foram feitas análises por imuno-histoquímica e também por proteômica baseada em alvos ou dirigida – onde sabíamos exatamente quais proteínas precisávamos quantificar”, disse Paes Leme.
A proteômica é o estudo de um conjunto de proteínas em uma amostra, seja em tecido ou célula, por exemplo, onde é possível identificar, quantificar, determinar modificações, localizar, avaliar atividade e interações de proteínas.
Bioinformática
Na primeira fase, os pesquisadores mapearam por microdissecção a laser e proteômica as proteínas no tecido de câncer de boca e correlacionaram com as características clínicas dos pacientes. Essa avaliação permitiu a identificação de várias proteínas, tais como CSTB, NDRG1, LTA4H, PGK1, COL6A1, ITGAV e MB, com padrões de abundância distintos dependendo da área do tumor avaliada e associação com importantes desfechos clínicos.
Na segunda fase, após identificar e quantificar as proteínas nas 120 amostras de tecido tumoral, os pesquisadores utilizaram duas estratégias para a verificação das proteínas.
“Em uma estratégia, avaliamos a abundância das proteínas selecionadas em amostras independentes de tecido de pacientes utilizando anticorpos por meio da imuno-histoquímica. Outra estratégia foi utilizar a saliva de pacientes, na qual monitoramos esses mesmos alvos pré-selecionados”, disse Paes Leme à Agência FAPESP.
Ela explica que o fluido foi escolhido uma vez que a lesão de câncer está localizada na boca, onde as células neoplásicas poderiam secretar proteínas.
“A saliva é uma fonte promissora de marcadores, além de ser um fluido obtido por meio de coleta não invasiva. Para tanto, foram verificadas as proteínas na saliva de 40 pacientes e, para obter maior confiabilidade do resultado nessa fase do estudo, as análises foram feitas em triplicatas técnicas”, disse.
Após a análise em amostras de saliva de pacientes, os pesquisadores utilizaram técnicas de bioinformática e de aprendizado de máquina para chegar à assinatura de prognóstico – verificar quais as proteínas ou peptídeos selecionados na primeira fase poderiam separar os pacientes com e sem metástase em linfonodo cervical.
“Além disso, também tínhamos a valiosa informação sobre a evolução clínica dos pacientes que participaram de forma voluntária do estudo, por meio da doação das amostras de saliva”, disse Paes Leme.
A partir desse resultado foi possível definir a assinatura de três peptídeos específicos de LTA4H, COL6A1 e CSTB, capazes de classificar os pacientes com e sem metástase em linfonodos cervicais, com grande potencial de ajudar os clínicos a superar as limitações dos exames e guiar as estratégias de tratamento personalizado.
A equipe de cientistas está dando andamento a uma nova pesquisa que tem por objetivo atuar de forma translacional e acessível na construção de biossensores para detectar essa assinatura de prognóstico na saliva de pacientes. Atualmente, os peptídeos podem ser identificados e quantificados por análise de espectrometria de massas e proteômica, técnicas custosas e incomuns em clínicas e hospitais.
“Queremos desenvolver um método mais simples, barato e acessível para profissionais da saúde para avaliar a progressão da doença a partir de testes que poderão ser feitos no consultório odontológico, consultório médico ou em laboratórios clínicos. No trabalho que acabamos de publicar, foi possível identificar essa assinatura de prognóstico por espectrometria de massas. Nossa ideia é desenvolver um biossensor focado na utilização dessa assinatura de prognóstico, para que tenha uso clínico, e orientar a definição do tratamento”, disse Paes Leme.
O artigo Combining discovery and targeted proteomics reveals a prognostic signature in oral cancer (doi: 10.1038/s41467-018-05696-2), de Carolina Moretto Carnielli, Carolina Carneiro Soares Macedo, Tatiane De Rossi, Daniela Campos Granato, César Rivera, Romênia Ramos Domingues, Bianca Alves Pauletti, Sami Yokoo, Henry Heberle, Ariane Fidelis Busso-Lopes, Nilva Karla Cervigne, Iris Sawazaki-Calone, Gabriela Vaz Meirelles, Fábio Albuquerque Marchi, Guilherme Pimentel Telles, Rosane Minghim, Ana Carolina Prado Ribeiro, Thaís Bianca Brandão, Gilberto de Castro Jr, Wilfredo Alejandro González-Arriagada, Alexandre Gomes, Fabio Penteado, Alan Roger Santos-Silva, Márcio Ajudarte Lopes, Priscila Campioni Rodrigues, Elias Sundquist, Tuula Salo, Sabrina Daniela da Silva, Moulay A. Alaoui-Jamali, Edgard Graner, Jay W. Fox, Ricardo Della Coletta e Adriana Franco Paes Leme, pode ser lido em www.nature.com/articles/s41467-018-05696-2.
Republicar
A Agência FAPESP licencia notícias via Creative Commons (CC-BY-NC-ND) para que possam ser republicadas gratuitamente e de forma simples por outros veículos digitais ou impressos. A Agência FAPESP deve ser creditada como a fonte do conteúdo que está sendo republicado e o nome do repórter (quando houver) deve ser atribuído. O uso do botão HMTL abaixo permite o atendimento a essas normas, detalhadas na Política de Republicação Digital FAPESP.





