

Científicos de Brasil, Alemania y Finlandia aplicaron esta técnica al estudio de una de las principales proteínas que actúan en el ciclo reproductor del SARS-CoV-2: la Mpro (En sentido horario, ilustración de la estructura dímera de la proteasa Mpro del SARS-CoV-2, simulación computarizada de la interacción del inhibidor N1 con Mpro, y representación bidimensional de la molécula de N1, con relieve para las interacciones con los sitios activos de Mpro/Journal of Biomolecular Structure and
Científicos de Brasil, Alemania y Finlandia aplicaron esta técnica al estudio de una de las principales proteínas que actúan en el ciclo reproductor del SARS-CoV-2: la Mpro
Científicos de Brasil, Alemania y Finlandia aplicaron esta técnica al estudio de una de las principales proteínas que actúan en el ciclo reproductor del SARS-CoV-2: la Mpro
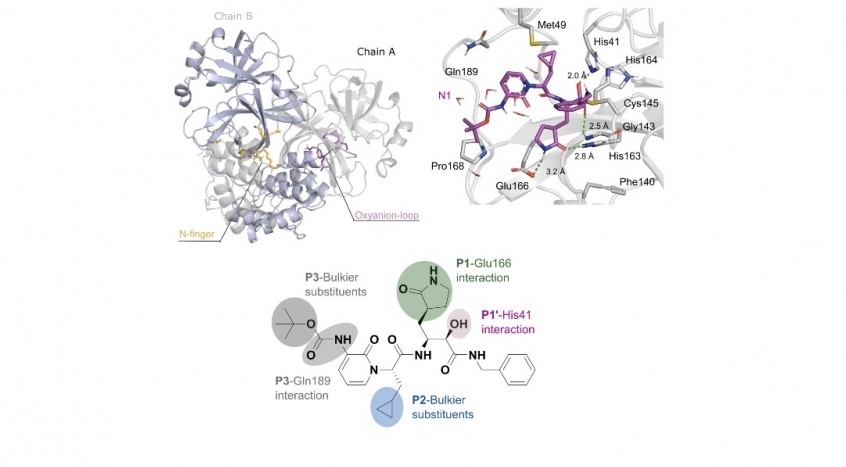
Científicos de Brasil, Alemania y Finlandia aplicaron esta técnica al estudio de una de las principales proteínas que actúan en el ciclo reproductor del SARS-CoV-2: la Mpro (En sentido horario, ilustración de la estructura dímera de la proteasa Mpro del SARS-CoV-2, simulación computarizada de la interacción del inhibidor N1 con Mpro, y representación bidimensional de la molécula de N1, con relieve para las interacciones con los sitios activos de Mpro/Journal of Biomolecular Structure and
Por Janaína Simões | Agência FAPESP – Un estudio a cargo de investigadores de Brasil, Alemania y Finlandia propone un nuevo modelo de simulaciones mediante el empleo de técnicas computacionales que promete acelerar la búsqueda de compuestos bioactivos contra el virus causante del COVID-19. Este procedimiento se aplicó en el estudio de una de las principales proteínas actuantes en el ciclo reproductor del SARS-CoV-2, que ha sido objeto de gran atención por parte de los científicos y de la industria farmacéutica, pues se la considera un blanco para drogas antivirales. Los investigadores estiman que el método que desarrollaron puede acortar el tiempo que requiere la fase inicial de la investigación básica, que se extiende entre dos y tres años, a menos de un año.
Tal como lo explican los autores, el SARS-CoV-2 posee una capa exterior de proteínas repletas del aminoácido cisteína, que debe estar intacta y activa para que el virus mantenga su actividad. La principal proteasa, la enzima que rompe las uniones peptídicas entre aminoácidos y proteínas, es la Mpro (del inglés main protease), encargada de dividir poliproteínas (cadenas proteicas) en proteínas menores. Estas, a su vez, están relacionadas con la producción del ARN que codifica a las proteínas estructurales del patógeno, como la spike (que se une al receptor de las células humanas para viabilizar la infección) y las que forman la envoltura vírica (la capa exterior que protege el material genético).
Investigadores y la industria de fármacos dirigen su mirada hacia Mpro como blanco para el desarrollo de medicamentos contra el COVID-19, porque la posibilidad de influir sobre, o bloquear esta división, constituye un quiebre de una de las etapas iniciales del ciclo de multiplicación del virus. Uno de los caminos es la síntesis de componentes químicos proyectados para unirse a puntos específicos de la proteína, de manera tal de bloquear este proceso e inactivarla.
Para desarrollar nuevos medicamentos, la industria farmacéutica tarda en promedio 20 años. Uno de los resultados del estudio publicado en el Journal of Biomolecular Structure and Dynamics fue la construcción de un patrón para la realización de simulaciones que puede reducir a menos de un año el tiempo destinado a la búsqueda de nuevos compuestos bioactivos contra el SARS-CoV-2. La búsqueda del compuesto activo se lleva a cabo al comienzo del desarrollo del medicamento, una etapa que normalmente tarda entre dos y tres años.
“Para producir las vacunas, se usó información conocida del SARS-CoV-1, del cual el SARS-CoV-2 es una evolución, lo cual aceleró la fase preclínica. No podemos hacer lo mismo en la investigación básica para desarrollar medicamentos pues no contamos con las informaciones de base necesarias a tal fin”, explica Glaucio Monteiro Ferreira, docente del Departamento de Análisis Clínicos y Toxicológicos de la Facultad de Ciencias Farmacéuticas de la Universidad de São Paulo (FCF-USP), en Brasil, y autor principal del artículo. Monteiro Ferreira condujo parte de la investigación en el Departamento de Oncología y Neumonología − Medicina Interna VIII del Hospital Universitario de Tubinga, en Alemania, con el apoyo de la FAPESP (16/12899-6 y 19/24112-9).
Otro factor que requiere más tiempo para la investigación y el desarrollo de un nuevo fármaco lo constituyen los estudios sobre su forma de administración: si por vía oral, en forma de pastillas o líquido, o en su forma inyectable, por ejemplo. “De tratarse de un medicamento elaborado para su ingestión, necesitamos asegurarnos de que pase todas las barreras del cuerpo humano para llegar adonde debe hacer efecto. Son detalles que requieren más tiempo para desarrollar un fármaco en comparación con una vacuna”, culmina.
Datos inéditos
Utilizando una combinación de herramientas tales como la bioinformática y la biología estructural, Monteiro Ferreira investigó la Mpro (también llamada 3CLpro), una de las proteínas virales que deben tener el aminoácido cisteína como sustrato o “alimento” para cumplir su función.
Diversos estudios anteriores muestran que existen proteínas que funcionan en el estado monomérico: son aquellas que tienen tan solo una cadena de proteínas. “Pero sabíamos que el SARS-CoV-1 tiene la cadena de proteínas duplicada: se trata de dímeros. Esto complica la búsqueda de un fármaco, pues debemos encontrar un compuesto que logre impedir que se forme la cadena doble”, comenta.
Un estudio anterior para el SARS-CoV-1 ya había arribado a resultados prometedores utilizando inhibidores covalentes unidos a la cisteína; es decir, un compuesto que se unía de manera más “fuerte” a este aminoácido. La intención de ese estudio era encontrar un medio de impedir que la cisteína quedase disponible para que la proteína se “alimentase” de ella, inhibiendo su acción.
El SARS-CoV-2 también es un dímero. Para iniciar la búsqueda de un compuesto que pueda interferir en la formación de las dos cadenas, entran entonces en escena las simulaciones. Mediante la aplicación de la dinámica molecular, un método de simulación computacional que estudia el movimiento físico de los átomos y las moléculas, se simuló un ataque del virus en una célula humana.
El equipo de científicos integrado por Monteiro Ferreira empezó a realizar simulaciones en las cuales la proteína tenía uno y dos ligandos. De este modo, se descubrió que las investigaciones enfocadas en el análisis de las proteínas con una cadena utilizaban un experimento, la difracción de rayos X, que captaba resultados después de que la proteína ya había consumido la cisteína y que se había separado. De este modo, no lograban enfocarse en el proceso de interés, el bloqueo de esa fase en la replicación de la proteína.
Como forma de aplicar la simulación para el estudio del potencial de nuevos fármacos y combatir al virus del COVID-19, se utilizaron dos inhibidores, el N1 y el N3, con efectos sobre la acción de la proteína Mpro. Los investigadores observaron que el primero es más eficaz, al no permitir la donación de carga eléctrica a la cisteína, es decir, al bloquear la “alimentación” de la proteína.
“Con las simulaciones que realizamos, llegamos más rápidamente a ese inhibidor, que puede efectivamente acabar con la enzima. Nuestras simulaciones indican el potencial de que ese compuesto se convierta en un medicamento mucho más potente y que esté mucho más cerca de transformarse en un fármaco”, apunta. Coincidentemente, Pfizer dio a conocer recientemente una iniciativa de búsqueda de un medicamento para el SARS-CoV-2 cuyo blanco es precisamente un inhibidor de la proteína Mpro, cosa que sucedió mucho después de que empezara la investigación de Monteiro Ferreira.
El investigador brasileño trabajó junto con Thales Kronenberger y Antti Poso, quienes se desempeñan en el Departamento de Oncología y Neumonología − Medicina Interna VIII, del Hospital Universitario de Tubinga, en Alemania, y en la Escuela de Farmacia de la Facultad de Ciencias de la Salud de la Universidad de Finlandia Oriental, en Finlandia, con Arun Kumar Tonduru, del mismo departamento en el Hospital Universitario de Tubinga, y con Rosario Dominguez Crespo Hirata y Mario Hiroyuki Hirata, ambos del Departamento de Análisis Clínicos y Toxicológicos de la Facultad de Ciencias Farmacéuticas de la USP. La investigación se realizó durante el posdoctorado de Monteiro Ferreira en el Laboratorio de Biología Molecular para el Diagnóstico y la Farmacogenómica (LBMAD), bajo la supervisión del profesor Hiroyuki Hirata.
Todos ellos firman el artículo intitulado SARS-COV-2 Mpro conformational changes induced by covalently bound ligands, que puede leerse en el siguiente enlace: www.tandfonline.com/doi/full/10.1080/07391102.2021.1970626.
Mon
Republicar
The Agency FAPESP licenses news via Creative Commons (CC-BY-NC-ND) so that they can be republished free of charge and in a simple way by other digital or printed vehicles. Agência FAPESP must be credited as the source of the content being republished and the name of the reporter (if any) must be attributed. Using the HMTL button below allows compliance with these rules, detailed in Digital Republishing Policy FAPESP.





