

Es un estudio realizado en Brasil en el cual se mapea el conjunto de genes que se activa cuando se infecta a ratones resistentes a Leishmania. Las moléculas que componen la vía de señalización responsable del combate contra el parásito pueden servir como biomarcadores para la realización de pronósticos de la enfermedad (esquema sencillo de la metodología y los resultados del experimento/ Juliana Ide Aoki)
Es un estudio realizado en Brasil en el cual se mapea el conjunto de genes que se activa cuando se infecta a ratones resistentes a Leishmania. Las moléculas que componen la vía de señalización responsable del combate contra el parásito pueden servir como biomarcadores para la realización de pronósticos de la enfermedad
Es un estudio realizado en Brasil en el cual se mapea el conjunto de genes que se activa cuando se infecta a ratones resistentes a Leishmania. Las moléculas que componen la vía de señalización responsable del combate contra el parásito pueden servir como biomarcadores para la realización de pronósticos de la enfermedad
Es un estudio realizado en Brasil en el cual se mapea el conjunto de genes que se activa cuando se infecta a ratones resistentes a Leishmania. Las moléculas que componen la vía de señalización responsable del combate contra el parásito pueden servir como biomarcadores para la realización de pronósticos de la enfermedad (esquema sencillo de la metodología y los resultados del experimento/ Juliana Ide Aoki)
Por Janaína Simões | Agência FAPESP – En un artículo publicado en la revista Scientific Reports, científicos brasileños vinculados a la Universidad de São Paulo (USP) describieron en ratones de qué manera ciertos factores genéticos pueden determinar si un individuo es susceptible o resistente a la leishmaniasis. Según los autores, estos resultados pueden ayudar a entender por qué también entre humanos tan solo algunos de los infectados desarrollan la enfermedad.
La leishmaniasis es causada por protozoos del género Leishmania que, según su especie, causan ulceraciones en la piel (leishmaniasis cutánea) o lesiones en órganos tales como el hígado y el bazo (leishmaniasis visceral). Los parásitos llegan a los humanos y a otros mamíferos a través de las picaduras de insectos. No existe una vacuna disponible y el tratamiento es largo, caro y complicado.
“Al observar la regulación de los genes en nuestro modelo de estudio, apuntamos a entender cuál es la estrategia que un organismo resistente la infección implementa y a determinar qué diferencias existen con relación a las de los organismos sensibles”, comentó Lucile Maria Floeter-Winter, coordinadora de la investigación en el Laboratorio de Fisiología del Instituto de Biociencias de la USP (IB-USP).
Esta investigación se llevó a cabo en el marco de dos Proyectos Temáticos apoyados por la FAPESP: “Estudios bioquímicos, moleculares y funcionales de la relación Leishmania-macrófago” y “La relación Leishmania-huésped desde el punto de vista de las ‘ómicas’”.
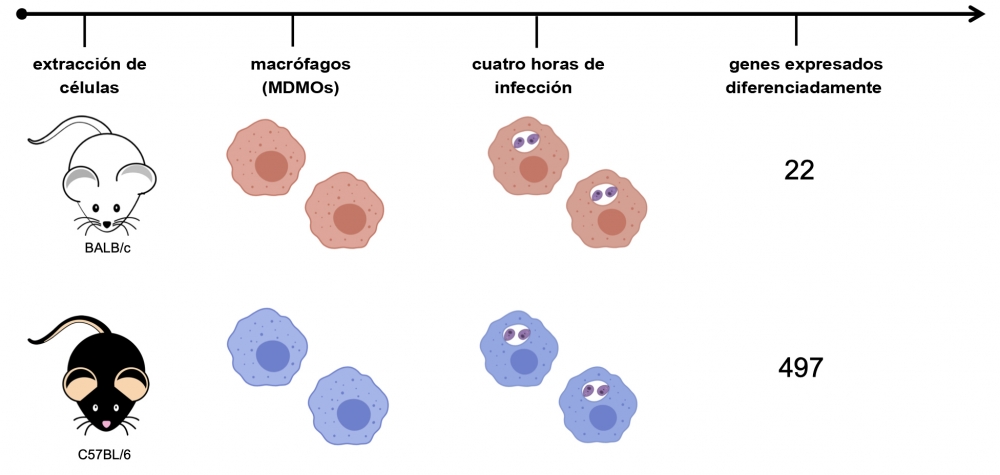
Los test se efectuaron con dos linajes de ratones: el linaje BALB/c, naturalmente sensible al parásito, y el linaje C57BL/6, naturalmente resistente. Ambos fueron infectados con la especie Leishmania amazonensis, causante de la forma cutánea de la enfermedad. A juicio de los investigadores, podrán efectuarse pronósticos de la enfermedad cuando se cuente con marcadores moleculares que indiquen la capacidad de resistir o no resistir a la infección.
El mapeo de la expresión génica
Los parásitos del género Leishmania operan con la estrategia de infección precisamente de las células que deberían combatirlos: los macrófagos. En esas células, los mismos se reproducen para luego infectar a otros macrófagos del organismo huésped.
Cuando los parásitos infectan a los macrófagos, pueden ocurrir dos cosas: o los macrófagos explotan y liberan parásitos, que entonces infectan a otros macrófagos situados a su alrededor, caso en el cual se considera que la infección está establecida, o los parásitos mueren debido a la acción de los macrófagos y la enfermedad no avanza.
Para diseñar un modelo experimental de la infección, los investigadores extrajeron células de la medula ósea de los ratones, diferenciaron esas células en macrófagos y las infectaron con parásitos. En este estudio se consideraron únicamente las primeras cuatro horas posteriores a la infección, cuando se emplearon los macrófagos infectados para la extracción de ARN y su posterior secuenciación. El mapeo de la expresión de los genes, realizado mediante el análisis del transcriptoma de ambos linajes de ratones, permitió verificar qué genes estaban expresándose durante las primeras cuatro horas de infección en cada grupo.
“Enfocamos nuestra atención al comienzo de la infección, cuando se disparan los mecanismos de respuesta”, explicó la investigadora del IB-USP, Juliana Ide Aoki, autora principal del artículo y becaria posdoctoral de la FAPESP.
“Al ser infectado, el organismo dispara un aviso para que los macrófagos produzcan un conjunto de moléculas encargadas de la respuesta a la infección. Cuando analizamos el transcriptoma, logramos ver qué segmentos del genoma han comprendido esa señal y han concretado una respuesta tendiente a combatir a los parásitos”, dijo Floeter-Winter.
En el conjunto de los genes expresados de los macrófagos estudiados en cultivo, se identificó un total de 12.641 genes. Con todo, en el BALB/c (sensible), tan solo un conjunto de 22 genes apareció con su expresión modificada, como una respuesta de combate contra el parásito. Una cifra mucho menor que los 497 observados en los ratones C57BL/6 (resistente).
“Este es el gran hallazgo de esta investigación. Tenemos un organismo que activa poquísimos genes y no es capaz de contener la infección, y, por otro lado, uno que acciona muchos genes que disparan la producción de moléculas con miras a controlar la infección y logra alcanzar ese objetivo”, comentó Floeter-Winter.
“Nuestros resultados muestran que el desarrollo de la enfermedad depende efectivamente de la genética del huésped y no solo del parásito, y puede explicar por qué existen pacientes que desarrollan la infección, en tanto que otros se muestran resistentes”, dijo.
De acuerdo con la investigadora, los ratones fueron tratados de la misma manera y se les dio la misma alimentación, lo cual descarta la influencia de factores ambientales en los resultados. Permanece como tema para investigaciones futuras develar por qué los animales C57BL/6 activan más genes para combatir la infección.
La determinación de las moléculas presentes en un organismo infectado que es capaz de controlar la infección permite sugerir marcadores para la evaluación y el pronóstico de la enfermedad en pacientes humanos. “Sería posible, por ejemplo, ver qué moléculas está expresando el paciente infectado y establecer un pronóstico con respecto a si desarrollará una enfermedad más prolongada, si desarrollará una infección con potencial más drástico o si produce moléculas que combaten la infección”, dijo la investigadora.
Asimismo, el aporte de este estudio a la Ciencia puede extrapolarse a otros aspectos de esta enfermedad. “Al entender de qué manera las Leishmanias logran establecer la infección, describimos mecanismos de respuesta que pueden utilizarse en otras enfermedades infecciosas, suministrando así información a otros científicos para la investigación en otros modelos”, culmina.
Este estudio recientemente publicado forma parte de un conjunto de investigaciones coordinadas por Floeter-Winter y tendientes a entender la relación entre huéspedes y parásitos, como la que identificó potenciales blancos para el tratamiento de la leishmaniasis (lea más en: agencia.fapesp.br/26323).
Puede leerse el artículo intitulado Differential immune response modulation in early Leishmania amazonensis infection of BALB/c and C57BL/6 macrophages based on transcriptome profiles (doi: 10.1038/s41598-019-56305-1), de Juliana Ide Aoki, Sandra Marcia Muxel, Ricardo Andrade Zampieri, Karl Erik Müller, Audun Helge Nerland y Lucile Maria Floeter-Winter, en el siguiente enlace: www.nature.com/articles/s41598-019-56305-1.
Republicar
The Agency FAPESP licenses news via Creative Commons (CC-BY-NC-ND) so that they can be republished free of charge and in a simple way by other digital or printed vehicles. Agência FAPESP must be credited as the source of the content being republished and the name of the reporter (if any) must be attributed. Using the HMTL button below allows compliance with these rules, detailed in Digital Republishing Policy FAPESP.





