

La metodología, descrita en la revista Nature Communications, permite entender reacciones de enzimas fundamentales en procesos biológicos tales como la fotosíntesis y la respiración celular (imagen: divulgación)
La metodología, descrita en la revista Nature Communications, permite entender reacciones de enzimas fundamentales en procesos biológicos tales como la fotosíntesis y la respiración celular
La metodología, descrita en la revista Nature Communications, permite entender reacciones de enzimas fundamentales en procesos biológicos tales como la fotosíntesis y la respiración celular
La metodología, descrita en la revista Nature Communications, permite entender reacciones de enzimas fundamentales en procesos biológicos tales como la fotosíntesis y la respiración celular (imagen: divulgación)
Por Karina Toledo
Agência FAPESP – En un artículo publicado recientemente en la revista Nature Communications, científicos de Brasil y Canadá describieron una nueva metodología destinada al estudio de la mecánica de las reacciones químicas catalizadas por metaloproteínas, un tipo de enzimas que contienen iones metálicos enlazados en la cadena polipeptídica.
Este trabajo cuenta con el apoyo de la FAPESP en el marco del proyecto intitulado “Desarrollo y aplicación de una simulación computacional y análisis espectroscópico para el estudio de metaloenzimas y proteínas flexibles”, coordinado por el profesor del Instituto de Química de la Universidad de São Paulo (IQ-USP) Guilherme Menegon Arantes, coautor del artículo.
“Las metaloproteínas –particularmente aquéllas que contienen átomos de hierro y de azufre– están implicadas en diversos procesos biológicos importantes para la vida, tales como la fotosíntesis y la respiración celular. Si logramos entender cómo se concretan la formación y la rotura de enlaces químicos en esas enzimas, comprenderemos mejor los procesos biológicos en los cuales las mismas participan”, dijo Arantes en entrevista a Agência FAPESP.
Como los átomos metálicos poseen una estructura electrónica más flexible, según explicó el investigador, son más reactivos que las moléculas puramente orgánicas, formadas únicamente por aminoácidos. Por tal motivo, las metaloproteínas son capaces de catalizar reacciones químicas sumamente difíciles, que no se concretarían sin la presencia de esas enzimas.
De acuerdo con Arantes, el gran desafío de los científicos interesados en estudiar estas proteínas radica en llegar a esos grupos metálicos en laboratorio. Al igual que cualquier proteína, para que estas enzimas asuman su forma funcional, las cadenas polipeptídicas se doblan con un origami al mismo tiempo en que se enrollan, en un proceso conocido como enmarañamiento. Como los grupos metálicos son altamente reactivos, se cree que el proceso evolutivo habría llevado a que éstos permaneciesen escondidos en el interior de la estructura tridimensional, de manera tal de evitar que ocurran reacciones indeseables.
“Esto facilita la actividad de la enzima, pero dificulta la vida del investigador que pretende estudiarla. Los métodos existentes hasta ahora consistían en desmontar la estructura tridimensional mediante el uso de condiciones físicas drásticas o de sustancias químicas. En nuestro trabajo se empleó una metodología que permite desenmarañar sólo parcialmente a la proteína sin dañar a la cadena polipeptídica, lo que permite estudiar las enzimas en una situación más análoga a las condiciones halladas en los organismos vivos”, explicó Arantes.
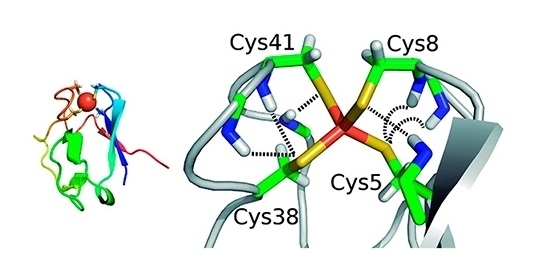
El grupo de la USP y de la University of British Columbia, en Canadá, está interesado particularmente en las proteínas provistas con centros de hierro-azufre. Las mismas están presentes en todos los organismos vivos y desempeñan funciones diversas, tales como la transferencia de electrones entre otras proteínas, la catálisis enzimática y la regulación genética. En el artículo recientemente publicado, se utilizó como modelo una proteína llamada rubredoxina, que efectúa la transferencia de electrones en microorganismos.
Mediante el empleo de una técnica de microscopía de fuerza atómica, los científicos de Canadá logran manipular la cadena polipeptídica que compone la proteína, estirándola y, al mismo tiempo, midiendo el desplazamiento y la fuerza necesaria para que se produzca el desenmarañamiento.
Simultáneamente, en Brasil, el grupo coordinado por Arantes realiza la simulación computacional de la reacción química. “Aunque en el experimento se logra medir con bastante precisión la fuerza y el desplazamiento necesario para el desenmarañamiento, esto no brinda una visión microscópica del proceso. En la simulación generamos una especie de película minuciosa, que muestra la estructura de la proteína y cómo se concreta la rotura o la formación de enlaces químicos. Por eso podemos comparar y validar la simulación con los datos reales obtenidos en el experimento”, dijo Arantes.
Para efectuar la simulación, el grupo de la USP se vale de una técnica conocida como de potenciales híbridos, que le redituó el Premio Nobel de Química de 2013 al austríaco Martin Karplus (Universidad Harvard), al sudafricano Michael Levitt (Universidad Stanford) y al israelí Arieh Warshel (Universidad del Sur de California).
“Es una técnica con la cual he venido trabajando desde mi doctorado, con el apoyo de la FAPESP, y consiste en aliar la mecánica cuántica con la mecánica molecular para describir la rotura o la formación de enlaces químicos en proteínas o en otros sistemas moleculares complejos”, dijo Arantes.
Si bien la mecánica cuántica es la teoría más apropiada para describir ese tipo de fenómenos, dijo el investigador, su uso se vuelve inviable cuando el sistema que se estudiará es tan complejo como una proteína, compuesta por miles de átomos.
“Ni siquiera las supercomputadoras son capaces de hacer operaciones de esa magnitud, por eso nos valemos de aproximaciones. La mecánica cuántica se emplea únicamente para estudiar el sitio catalítico de la enzima, y todo el resto de la molécula que no está implicado en la reacción química se describe a través de la mecánica molecular, sin detallar la estructura electrónica”, explicó Arantes.
En un trabajo anterior, publicado en la revista Angewandte Chemie, el grupo simuló cómo se produce la rotura de la unión hierro-azufre en la rubredoxina durante el desenmarañamiento sin presencia de cualquier otro reactivo químico.
En el artículo más reciente, se informa el agregado de un agente nucleofílico –que se une al átomo de hierro– y un agente electrofílico, que se une al átomo de azufre.
“El grupo experimental midió la alteración de la fuerza necesaria como para romper la unión natural del hierro con el azufre en presencia de otros agentes competitivos. Hicimos la simulación para entender cuál es el mecanismo de la reacción química y cómo la misma se ve facilitada debido al desenmarañado parcial de la proteína y por la presencia de los agentes químicos”, comentó Arantes.
A juicio del investigador, la técnica combinada puede constituirse como una herramienta poderosa para estudiar la reacción y la estabilidad de diversas metaloproteínas.
“Al entender mejor la reactividad de esos centros metálicos, podemos intentar hallar medios para controlar los escapes de electrones, que son responsables de formar alrededor del 90% de los radicales libres generados en el interior de las células. Estos radicales libres reaccionan con biomoléculas y pueden perjudicar el funcionamiento celular”, afirmó Arantes.
Republicar
The Agency FAPESP licenses news via Creative Commons (CC-BY-NC-ND) so that they can be republished free of charge and in a simple way by other digital or printed vehicles. Agência FAPESP must be credited as the source of the content being republished and the name of the reporter (if any) must be attributed. Using the HMTL button below allows compliance with these rules, detailed in Digital Republishing Policy FAPESP.





